1286 - CAMBIOS EN LA METILACIÓN DEL ADN DURANTE LA SEPSIS: DESCUBRIENDO LOS MECANISMOS EPIGENÉTICOS DE LA DESREGULACIÓN INMUNITARIA
1Hospital Universitario Dr. Peset, Valencia, España. 2Universitat de València, Valencia, España. 3Consorcio Centro de Investigación Biomédica en Red del Instituto de Salud Carlos III (CIBER-ISCIII), Valencia, España. 4Instituto de Investigación Sanitaria INCLIVA, Valencia, España.
Objetivos: La sepsis se define por una respuesta inmunológica desregulada ante una infección, que puede llevar al fallo de órganos y la muerte (Singer, 2016). Sin embargo, sus mecanismos siguen sin comprenderse completamente. La epigenética estudia los cambios que regulan la expresión génica sin modificar la secuencia del ADN, en respuesta a estímulos externos. La metilación del ADN es uno de los principales cambios epigenéticos, y juega un papel clave en la regulación del sistema inmunitario (Cross, 2019). El objetivo de este estudio fue conocer los cambios en la metilación del ADN que ocurren durante la sepsis.
Métodos: Estudio prospectivo de cohortes de pacientes con sepsis de origen comunitario, que ingresaron en un hospital terciario entre septiembre de 2019 y diciembre de 2023. Se compararon con un grupo control de pacientes sin sepsis, con una infección del mismo foco, con el mismo sexo y edad ± 5 años. Se extrajeron muestras de sangre y se almacenaron en un biobanco en las primeras 24 h de ingreso. Se seleccionaron 16 pacientes con sepsis y 16 controles, y se realizó un ensayo mediante arrays de metilación EPIC850k 2.0 (Illumina) para analizar las diferencias en la metilación del ADN entre ambos grupos. El proyecto fue aprobado por el Comité Ético de Investigación y se financió parcialmente con una beca de ayuda a la investigación FEMI-2022. Se obtuvo consentimiento informado de todos los pacientes.
Resultados: Se obtuvieron 4,640 posiciones diferencialmente metiladas (DMP) significativas (FDR < 0,05). Se realizó un mapa de calor (fig. 1) y un gráfico de volcán (fig. 2) con los valores de metilación diferencial de estas DMP. En la tabla 1 se pueden consultar las 10 DMP significativas asociadas a genes con mayores diferencias en la metilación. A continuación, se realizó un análisis de sobrerrepresentación (ORA, over-representation analysis) cuyos resultados se muestran en la tabla 2.
|
Tabla 1. Top 10 DMPs significativas (FDR < 0,05) asociadas a genes con la mayor diferencia media de β-valor |
||||||
|
Sonda CpG |
FDR |
β-valor (Dif. media) |
Cromosoma |
Posición |
Genes |
|
|
% |
Sentido |
|||||
|
cg10833066_TC11 |
0,012 |
24,3 |
Hipermetilado |
CHR12 |
111369663 |
PHETA1 |
|
cg01022219_TC11 |
0,007 |
15,8 |
Hipometilado |
CHR18 |
13641736 |
LDLRAD4 |
|
cg05779786_BC11 |
0,049 |
15,5 |
Hipometilado |
CHR2 |
236120205 |
AGAP1 |
|
cg07110356_TC11 |
0,031 |
15,0 |
Hipometilado |
CHR17 |
58278070 |
MPO |
|
cg05101463_BC21 |
0,030 |
14,8 |
Hipometilado |
CHR5 |
43191091 |
NIM1K |
|
cg07252680_TC21 |
0,002 |
14,0 |
Hipometilado |
CHR14 |
94390887 |
SERPINA1 |
|
cg02694427_BC11 |
0,012 |
13,5 |
Hipometilado |
CHR2 |
176099784 |
HOXD12 |
|
cg07003993_BC21 |
0,011 |
13,5 |
Hipermetilado |
CHR11 |
67022607 |
SYT12 |
|
cg17525495_BC11 |
0,041 |
12,9 |
Hipometilado |
CHR17 |
58324373 |
TSPOAP1-AS1 |
|
cg01942530_BC21 |
0,032 |
12,3 |
Hipermetilado |
CHR8 |
142880098 |
CYP11B1 |
|
Tabla 2. Términos GO - Biological process significativos obtenidos en el análisis ORA a partir de las DMP significativas |
||
|
Términos enriquecidos |
Recuento de genes |
p |
|
Neutrophil mediated immunity (GO:0002446) |
66 |
< 0,001 |
|
Neutrophil degranulation (GO:0043312) |
65 |
< 0,001 |
|
Neutrophil activation involved in immune response (GO:0002283) |
65 |
< 0,001 |
|
Inflammatory response (GO:0006954) |
26 |
0,014 |
|
Regulation of immunoglobulin production (GO:0002637) |
8 |
0,014 |
|
Positive regulation of vascular associated smooth muscle cell proliferation (GO:1904707) |
7 |
0,014 |
|
Purinergic nucleotide receptor signaling pathway (GO:0035590) |
7 |
0,014 |
|
Positive regulation of monocyte differentiation (GO:0045657) |
5 |
0,018 |
|
Regulation of monocyte differentiation (GO:0045655) |
6 |
0,019 |
|
Positive regulation of T-helper 2 cell differentiation (GO:0045630) |
4 |
0,021 |
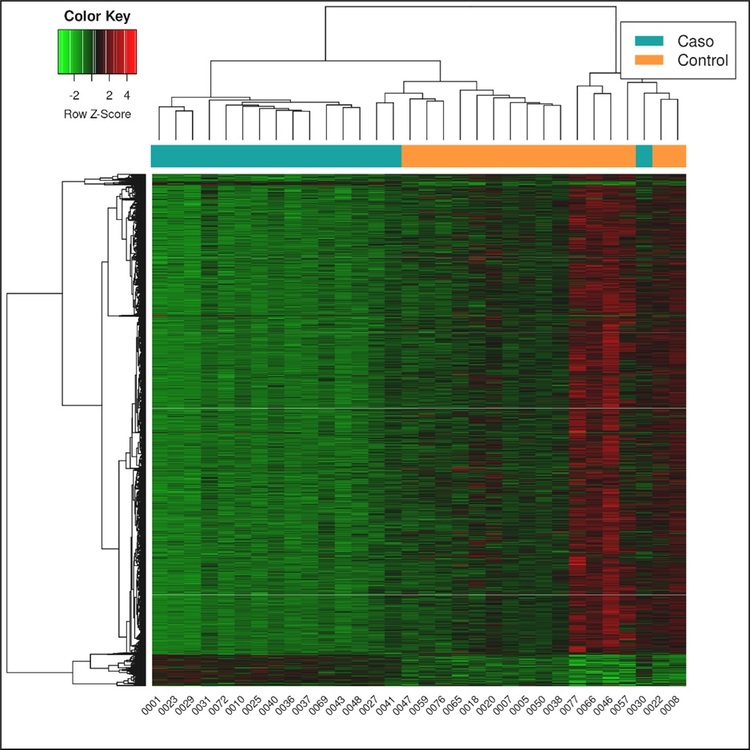
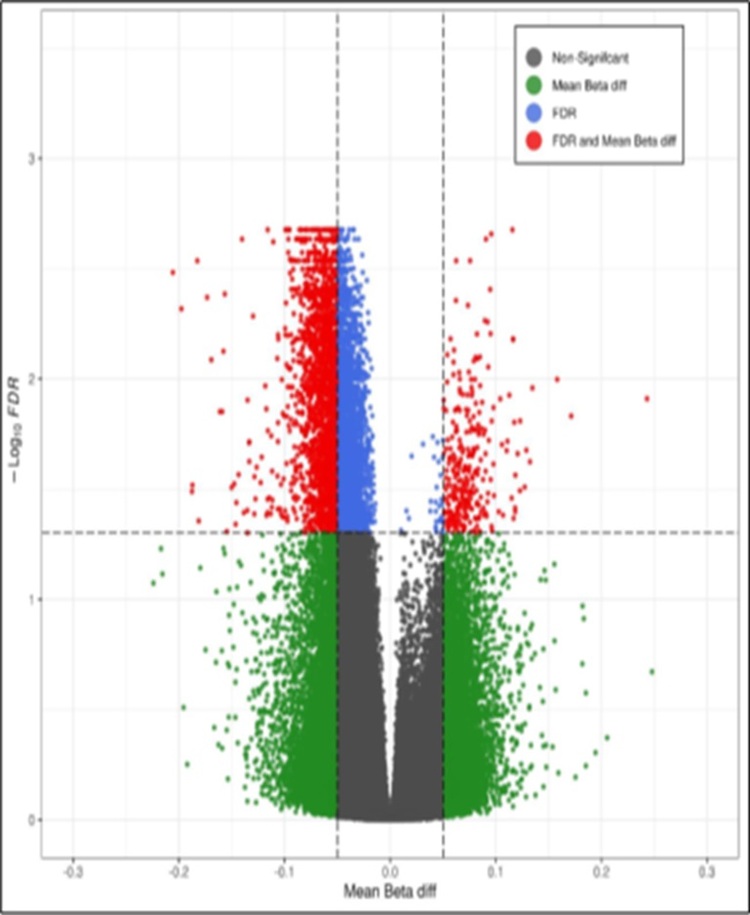
Discusión: El análisis ORA mostró que los cambios en la metilación afectan a procesos relacionados con la inmunidad, entre los que destacan la degranulación de neutrófilos, la diferenciación de monocitos y linfocitos Th2, y la producción de inmunoglobulinas; así como componentes de los gránulos azurófilos. Entre los genes identificados destacan: MPO: codifica la enzima mieloperoxidasa (MPO), cuyos niveles se relacionan con escalas de gravedad y la mortalidad en la sepsis (Schrijver, 2017). SERPINA1: codifica la proteína alfa-1 antitripsina (AAT), que aumenta en las infecciones para evitar el daño proteolítico de la elastasa de neutrófilos. Algunos estudios muestran utilidad como biomarcador en la sepsis (Zhou, 2021). Estos resultados sugieren patrones de metilación distintos entre pacientes con sepsis y sin sepsis, que parecen jugar un papel fundamental en las alteraciones en la regulación inmunitaria en la sepsis (Lorente-Sorrolla, 2019; Binnie 2020).
Conclusiones: Existen diferencias en la metilación del ADN entre los pacientes con sepsis y aquellos con infección que no desarrollan sepsis. Los genes SERPINA1 y MPO tienen implicaciones en la regulación del sistema inmunitario y están hipometilados durante la sepsis, por lo que podrían tener utilidad como biomarcadores en la sepsis.






