Un nuevo brote de coronavirus surgió el pasado 31 de diciembre de 2019 en Wuhan, China, causando conmoción entre la comunidad médica y el resto del mundo. Esta nueva especie de coronavirus fue denominada como 2019-nCoV, causante de un gran número de casos y fallecimientos en China y en cantidad creciente fuera de ella, convirtiéndose en una emergencia de salud pública a nivel mundial. 2019-nCoV es un virus con alta homología con otros coronavirus patogénicos, como los originados por zoonosis con murciélagos (SARS-CoV) causantes de aproximadamente 646 muertes en China a principios de la década. Su tasa de mortalidad no es tan elevada (aproximadamente del 2-3%), pero su rápida propagación ha propiciado la activación de protocolos para detener su diseminación. Este patógeno tiene el potencial para convertirse en pandemia, por lo que es vital seguir las recomendaciones de cuidado personal dictadas por la Organización Mundial de la Salud.
A new coronavirus outbreak emerged on the 31st of December 2019 in Wuhan, China, causing commotion among the medical community and the rest of the world. This new species of coronavirus has been termed 2019-nCoV and has caused a considerable number of cases of infection and deaths in China and, to a growing degree, beyond China, becoming a worldwide public health emergency. 2019-nCoV has high homology to other pathogenic coronaviruses, such as those originating from bat-related zoonosis (SARS-CoV), which caused approximately 646 deaths in China at the start of the decade. The mortality rate for 2019-nCoV is not as high (approximately 2-3%), but its rapid propagation has resulted in the activation of protocols to stop its spread. This pathogen has the potential to become a pandemic. It is therefore vital to follow the personal care recommendations issued by the World Health Organisation.
En diciembre de 2019 se identificaron en Wuhan (China) una serie de casos de neumonía originados por un nuevo coronavirus. Este nuevo coronavirus tiene distintas denominaciones: 2019-nCoV según la OMS y SARS-CoV-2 según el Comité Internacional de Taxonomia de Virus. La enfermedad que lo causa se ha denominado 2019-nCoV1. El 7 de enero de 2020, el nuevo coronavirus fue anunciado oficialmente por las autoridades chinas como el agente causal de dichas infecciones1.
Los coronavirus son virus envueltos de ARN de sentido positivo no segmentados que pertenecen a la familia Coronaviridae y al orden Nidovirales, y se distribuyen ampliamente en humanos y otros mamíferos, originando múltiples afecciones que van desde una gripe «común» hasta la muerte2.
Hasta el 13 de febrero de 2020 varios casos han sido registrados. Según la Organización Mundial para la Salud (OMS): se han reportado 46.997 casos a nivel global, de los cuales 46.550 (99,04%) han sido confirmados en China, y de esos han muerto 1.368 (2,93%), lo que la convierte en una enfermedad catalogada como emergencia de salud pública a nivel mundial3,4.
En el siguiente link se puede observar el mapa actualizado en tiempo real de la situación mundial de los casos del coronavirus con incidencia y localización: https://gisanddata.maps.arcgis.com/apps/opsdashboard/index.html#/bda7594740fd40299423467b48e9ecf6
Características estructurales de los coronavirusLos coronavirus se pueden diferenciar en 4 géneros: alfa, beta, delta y gamma, de los cuales hasta el momento se sabe que los coronavirus de tipo alfa y beta infectan a los humanos5, provocando enfermedades que van desde el resfriado común hasta afecciones más graves, como el síndrome respiratorio de Oriente Medio (MERS-CoV) y el síndrome respiratorio agudo severo (SARS-CoV), que causó miles de muertes en 20026,7. Cuatro de los HCoV (HCoV 229E, NL63, OC43 y HKU1) son endémicos en todo el mundo y representan del 10 al 30% de las infecciones del tracto respiratorio superior en adultos5.
Estructuralmente los coronavirus son virus esféricos o pleomórficos, cuyo diámetro varía de 80-120nm. Diversos análisis por microscopia electrónica han identificado la superficie del virión, descubriendo que son estructuras organizadas por proyecciones que a su vez están constituidas por trímeros de la glicoproteína viral S (Spike)8. Adicionalmente, se han identificado otras proyecciones cortas formadas por dímeros de las proteínas HE (Hemaglutinina-Esterasa), la cual se ha observado en algunos beta-coronavirus9. Por su parte, la envoltura viral se encuentra reforzada por la glicoproteína de Membrana (M) (la más abundante en la superficie del virión), que se encuentra embebida en la membrana por 3 dominios transmembranales10. Otro componente estructural del virión es la proteína de Envoltura (E), una proteína de un tamaño pequeño que es altamente hidrofóbica y que se encuentra en una proporción menor que las demás11. Las proteínas virales de los coronavirus se encuentran embebidas en una membrana lipídica que es originada de la célula infectada. Internamente, la partícula viral está constituida por una proteína adicional conocida como Nucleoproteína (N), la cual se une al ARN viral en una estructura helicoidal similar a una cuerda con cuentas, protegiendo así al ARN de su degradación (fig. 1A)8.
A) Partícula de coronavirus. Este tipo de virus contiene un material genético de ARN de hebra sencilla de polaridad positiva [(+) ssARN], con un tamaño de 27-32kilobases. Está constituido por una nucleocápside y esta a su vez está compuesta por (+) ssARN y la Nucleoproteína; esta estructura está cubierta de una bicapa lipídica. Aquí se encuentran otras proteínas estructurales del coronavirus como es la proteína Spike que cubre esta partícula viral, así como dímeros de Hemaglutinina-Esterasa (HE); también consta la proteína de Envoltura (E) altamente hidrofóbica y la proteína de Membrana (M), la más abundante en la superficie del virión. B) Organización de los genes en el genoma 2019-nCoV. Los genes estructurales se muestran de color rosa y los genes no estructurales de color azul.
El genoma de los coronavirus es de ARN de hebra sencilla no segmentado y de polaridad positiva (+ssARN), que cuenta con un tamaño de 27 a 32kilobases. El ARN genómico presenta modificaciones como poliadenilaciones en la región 3’ terminal; en cambio, la región 5’ terminal contiene una estructura tipo cap12. Dentro de este ARN existen múltiples marcos de lectura abiertos (6-11 ORFs). El primer ORF codifica para aproximadamente 16 proteínas no estructurales, mientras que los ORF restantes codifican para proteínas accesorias y no estructurales (fig. 1B)13.
Los análisis de las secuencias del genoma del 2019-nCoV en comparación con SARS-CoV son bastante similares, sin embargo, existen ciertas diferencias como la falta de la región codificante para la proteína 8a en 2019-nCoV, por lo que podría tener una implicación en una menor patogénesis en comparación con el SARS-CoV14.
OrigenUna incógnita que continúa siendo investigada es el reconocimiento del origen zoonótico de dicho virus, pero debido a su estrecha similitud con los coronavirus de murciélago, es probable que estos sean el reservorio primario del virus, pues con la reaparición de esta nueva clase de coronavirus se realizaron diversos estudios y se descubrió que el 2019-nCoV es un 96% idéntico a nivel del genoma a un coronavirus de murciélago; el mismo estudio reveló que dicho virus pertenece a la especie de SARS-CoV15. Es así como se especula que el SARS-CoV se transmitió a los humanos de animales exóticos en mercados en el brote de hace 18 años15, mientras que el MERS-CoV se transmitió de los camellos a los humanos16.
Los informes documentaron que muchos de los primeros pacientes identificados tenían como factor común el contacto con un mercado de mariscos y animales, sin embargo, otros no establecieron contacto con dicho lugar en ningún momento, lo que evidencia la infección limitada de persona a persona después de identificar grupos de casos entre familias, así como la transmisión de pacientes a trabajadores de la salud17. A su vez, un estudio publicado recientemente estimó que un 95% de los casos de infecciones por 2019-nCoV en Wuhan presentaron síntomas antes del 12 de enero de 202018,19, dato que asociado a su período de incubación sugiere una alta posibilidad de riesgos de propagación de la enfermedad relacionada con los viajes20,21.
TransmisiónGeneralmente los coronavirus se replican primordialmente en las células epiteliales del tracto respiratorio inferior y en menor medida en las células de las vías respiratorias superiores; es por eso que la transmisión ocurre principalmente de pacientes con enfermedad reconocida y no de pacientes con signos leves e inespecíficos, es decir, que se cree que la propagación ocurre solo después de que se presentan signos de enfermedad del tracto respiratorio inferior22,23. Sin embargo, los pacientes con infección por 2019-nCoV detectados en estado severo o fatal tienen una mayor probabilidad de transmitir este virus, ya que eliminan una mayor cantidad de partículas infectivas en comparación con los pacientes que presentan la infección de forma leve o asintomática. Identificar y poner en cuarentena a estos pacientes en instituciones de salud donde se han producido brotes, junto con la implementación de un control de infección adecuado, y los constantes informes sobre los casos en diferentes países, ha sido eficaz para reducir la transmisión y contener brotes de la enfermedad24.
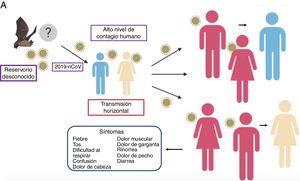
El número promedio de casos nuevos que genera un caso de coronavirus a lo largo de su período infeccioso (R0) oscila entre 2,24 (IC 95%: 1,96-2,55) y 3,58 (IC 95%: 2,89-4,39)21, es decir, que una persona puede llegar a infectar aproximadamente de 2 a 4 personas, lo que significa que la infección puede propagarse rápida y ampliamente entre la población (fig. 2).
Propagación del 2019-nCoV. Se tienen diferentes hipótesis sobre la transmisión de animal a humano, la más fuerte es que su origen proviene de murciélagos; mientras que la transmisión entre humanos se ha reportado por vía respiratoria, por lo que eleva más fácilmente el contagio entre las poblaciones. El cuadro sintomático es muy común con fiebre, tos, dolor muscular y problemas respiratorios, entre otros.
¿Quiénes son más susceptibles al contagio? El nuevo coronavirus puede infectar a personas de todas las edades, aunque las personas mayores y aquellas con afecciones médicas preexistentes (como asma, diabetes y enfermedades cardíacas) parecen ser más vulnerables a enfermarse seriamente con el virus, reportándose así una tasa de mortalidad > 8% en personas mayores a 70 años. Según informes, la mayoría de los pacientes fallecidos tenían una edad promedio de 56 años, y en gran parte padecían otras enfermedades (cardíacas, accidente cerebrovascular, diabetes, etc.) que pudieron haberlos hecho más vulnerables al virus. El Centro Chino para el Control y la Prevención de Enfermedades dijo que de 1 a 2 hombres estaban infectados por cada mujer. Se especula que la poca susceptibilidad de las mujeres a las infecciones virales puede deberse a la protección del cromosoma X «extra» con el que cuentan en comparación con los hombres25. Por su parte, los niños comprenden una población peculiar que posee un sistema inmune distinto al de los adultos, por lo que la transmisión del virus a través de sus madres con infección sospechada o confirmada ocurre fácilmente26. Sin embargo, se ha observado una severidad menor y una mortalidad extremadamente baja.
Cuadro clínicoEl diagnóstico de la enfermedad se ha hecho como una neumonía inducida por virus con base en los síntomas clínicos que observaron en los pacientes, similares a los de otros virus respiratorios, a su historia de exposición con otras personas con el virus, y la historia de visitas a zonas afectadas7,27. A su vez, un estudio detallado de los primeros 99 pacientes atendidos en el Hospital Wuhan Jinyintan, que se llevó a cabo del 1 al 20 de enero de 2020, arrojó que de los 99 pacientes con neumonía 2019-nCoV, el 49% tenían antecedentes de exposición al mercado de mariscos de Huanan y el 51% tenían enfermedades crónicas. La edad promedio de estos pacientes fue de 55,5 años, incluidos 67 hombres y 32 mujeres, y el período de incubación del virus se estimó en entre 7 y 14 días27. Los pacientes estudiados tenían manifestaciones clínicas de fiebre (83%), tos (82%), dificultad para respirar (31%), dolor muscular (11%), confusión (9%), dolor de cabeza (8%), dolor de garganta (5%), rinorrea (4%), dolor en el pecho (2%), diarrea (2%), náuseas y vómitos (1%). Según el examen por imágenes, el 75% de estos pacientes presentaron neumonía bilateral, el 14% mostró moteado múltiple y opacidad en vidrio esmerilado y el 1% tenía neumotórax. Igualmente, el 17% desarrolló síndrome de dificultad respiratoria aguda, y de ellos el 11% empeoraron en un corto período de tiempo y murieron por insuficiencia orgánica múltiple23,28. A su vez, los leucocitos estaban por debajo del rango normal en el 9% de los pacientes y por encima del rango normal en el 24%, y el 38% de ellos tenían neutrófilos por encima del rango normal. Por su parte, los linfocitos y la hemoglobina estuvieron por debajo del rango normal en muchos pacientes, y las plaquetas estaban por debajo del rango normal en el 12% de ellos y por encima del rango normal en el 4%; 43 de estos pacientes tenían diferentes grados de anormalidad de la función hepática, con alanina aminotransferasa (ALT) o aspartato aminotransferasa (AST) por encima del rango normal; y un paciente tenía daño grave de la función hepática28,29.
Clasificación de los pacientes de acuerdo con sus síntomas clínicosConsiderando los síntomas clínicos de los pacientes, ¿cómo se sabe cuando hablamos de un caso sospechoso, un caso probable o un caso confirmado de 2019-nCoV? Según la OMS: un caso sospechoso es un paciente con infección respiratoria aguda grave (fiebre, tos, y que requiere ingreso al hospital) y sin otra etiología que explique completamente la presentación clínica, y un historial de viaje o residencia en China durante los 14 días antes del inicio de los síntomas; o en su lugar, un paciente con alguna enfermedad respiratoria aguda y al menos uno de los siguientes puntos durante los 14 días previos al inicio de la aparición de los síntomas: a) contacto con un caso confirmado o probable de infección por 2019-nCoV, o si b) trabajó o asistió a un centro de atención médica donde pacientes con 2019-nCoV confirmado o probable y pacientes con enfermedad respiratoria aguda estaban siendo tratados. Por otra parte, un caso probable es un paciente cuyas pruebas para 2019-nCoV no son concluyentes o se da positivo usando un ensayo para coronavirus y sin evidencia de laboratorio de otros patógenos respiratorios. Finalmente, hablamos de un caso confirmado cuando el paciente tiene resultados de laboratorio positivos para la infección 2019-nCoV, independientemente de los signos y síntomas clínicos30.
Pruebas clínicas para el diagnósticoEntre las principales prioridades para facilitar las intervenciones de salud pública en los pacientes se encuentra el diagnóstico de laboratorio. En el caso de una infección respiratoria aguda, la RT-PCR se utiliza comúnmente para identificar virus causales de secreciones respiratorias30,31.
La prueba para la detección de la secuencia del gen de la envoltura viral ha sido implementada eficazmente por 35 laboratorios, sin embargo, el algoritmo diagnóstico emplea otras secuencias del genoma viral para confirmar positividad para 2019-nCoV al detectar secuancias del gen de la ARN polimerasa viral y la Nucleoproteína32.
Otras muestras a recolectar son:
- a)
Extracción de ARN de muestras clínicas con el sistema MagNA Pure 96.
- b)
Material respiratorio (nasofaríngeo e hisopo orofaríngeo en pacientes ambulatorios y esputo [si se presenta] y/o aspirado endotraqueal o lavado broncoalveolar en pacientes con enfermedad respiratoria severa)33.
- c)
Suero para pruebas serológicas, muestra aguda y muestra convaleciente (adicional a materiales respiratorios)33
Los objetivos de las pruebas de diagnóstico son detectar las causas frecuentes de neumonía temprana, para apoyar actividades de control de enfermedades y trabajar con laboratorios de referencia que pueden realizar la detección de pancoronavirus y secuenciación dirigida31.
Medidas de prevenciónLas recomendaciones estándar dictadas por la OMS para prevenir la propagación de la infección incluyen: lavarse las manos regularmente, especialmente después del contacto con personas enfermas o su entorno, cubrirse la boca y la nariz al toser y estornudar, cocinar bien la carne y los huevos, evitar el contacto cercano con cualquier persona que presente síntomas de enfermedades respiratorias (tos y estornudos), evitar viajar a las ciudades y áreas afectadas, y evitar el contacto cercano con animales vivos o muertos de granja o salvajes. En el caso de los viajeros con síntomas de infección respiratoria aguda, deben practicar la etiqueta de la tos (mantener la distancia, cubrirse la boca al toser y estornudar con pañuelos desechables o ropa y lavarse las manos adecuadamente)7,34,35.
Aquellas personas que hayan tenido contacto con casos de pacientes que hayan sido catalogados como probables y/o confirmados de 2019-nCoV deben ser monitorizados durante 14 días desde el último contacto que tuvieron con ellos sin protección y deben limitar los traslados a lugares fuera de su lugar de residencia para evitar una posible propagación.
Una de las medidas preventivas más utilizadas es el uso de cubrebocas, pero ¿pueden detener la propagación? Las mascarillas quirúrgicas para el público no son una protección 100% efectiva contra los virus o bacterias transportadas en el aire, dado que no cuentan con un filtro de aire adecuado y dejan los ojos expuestos, y aunque podrían ayudar a reducir el riesgo de contraer el virus mediante estornudos o tos ajenos, lo óptimo es el uso de los respiradores que tienen un filtro de aire especializado, ya que están diseñados específicamente para proteger a una persona contra partículas potencialmente peligrosas que se encuentren en el aire, es decir, las mascarillas FFP, de las cuales existen 3 tipos:
- •
Mascarilla FFP1: filtra aproximadamente el 78% de las partículas del aire, por lo que protege de residuos no tóxicos y no fibrogénicos de polvo o aerosoles, y previene la inhalación de residuos y olores molestos.
- •
Mascarilla FFP2: filtra aproximadamente el 92% de las partículas del aire, y protege de residuos no tóxicos y elementos fibrogénicos, evitando la inhalación de fluidos tóxicos de polvo, aerosoles y humos.
- •
Mascarilla FFP3: filtra el 98% de las partículas del aire, protegiéndonos frente a tipos venenosos y tóxicos de polvo, humo y aerosoles, así como bacterias, virus y esporas de hongos.
La OMS recomienda que para procedimientos de aislamiento o con posible generación de aerosoles infecciosos (tuberculosis, sarampión, varicela, SRAS…) se haga uso de un respirador con una eficiencia de filtración de al menos el 95% para partículas de 0,3μm de diámetro, lo que equivale a una mascarilla N95 según la normativa americana NIOSH, pero como la normativa americana no equivale a la europea, este nivel de protección para la población en general se encuentra entre la FFP2 y FFP3.
En general, el uso de máscaras solo se recomienda cuando existe exposición con pacientes con enfermedades respiratorias, como es hospitales y consultorios. Se deben seguir estrictamente los lineamientos de uso de las máscaras, cuando se expone la persona a pacientes infectados, pues la mala manipulación y contacto de las manos con los ojos y con el lado externo de la máscara aumentan los riesgos.
¿A quién notificar cuando existe sospecha de infección o casos confirmados?Cualquier persona que haya tenido contacto con enfermos y que cumpla las características clínicas de la enfermedad se convierte en un caso sospechoso y debe ser confirmado mediante pruebas de laboratorio30. La OMS solicita que las autoridades nacionales denuncien casos confirmados de nueva infección por coronavirus dentro de las 24h de identificación, proporcionando el conjunto mínimo de datos del paciente30. En el caso de México, ante la presencia de signos y síntomas de la enfermedad, puede comunicarse a la Unidad de Inteligencia Epidemiológica y Sanitaria (UIES), las 24h del día, los 7 días de la semana, al número 8000044800. En España deben comunicarse en Madrid con el Ministerio de Sanidad al número de teléfono 915961000 o al Instituto Nacional de Gestión Sanitaria, al 913380000.
TratamientoEntre tanto, numerosos laboratorios de investigación en la actualidad buscan un tratamiento que elimine la infección por el MERS-CoV, ya sea con medicamentos que ya formen parte de la industria farmacéutica y sean empleados para otras enfermedades, o bien buscando nuevas alternativas más específicas del virus, pues no se dispone actualmente de vacuna alguna ni de tratamiento específico, aunque hay varias vacunas y tratamientos específicos en fase de desarrollo.
A pesar del poco tiempo, se prevé que haya una vacuna lista para probar en animales aproximadamente en un mes, y en 3 meses podría estar lista para las personas. Por ahora, el tratamiento es únicamente de apoyo y depende del estado clínico del paciente36.
Los antivirales de amplio espectro, como el remdesivir, el lopinavir/ritonavir y el interferón beta, han demostrado ser prometedores contra el MERS-CoV en modelos animales y se está evaluando su actividad contra el actual 2019-nCoV36. Además, científicos del Centro de Investigación de Vacunas del Instituto Nacional de Alergias y Enfermedades Infecciosas de Washington DC han utilizado enfoques de plataformas de vacunas de ácido nucleico para contrarrestar la afección37.
ConclusiónLa OMS se encuentra colaborando con especialistas en salud pública y sanidad animal, clínicos y científicos internacionales, para reunir e intercambiar datos científicos que permitan conocer mejor el virus y la enfermedad que provoca; un ejemplo de estos datos son la identificación de las nuevas rutas de secreción en pacientes infectados. De igual manera, se han enfocado en determinar las prioridades de respuesta a los brotes, las estrategias terapéuticas de tratamiento y los enfoques de la atención clínica. Existen análisis que sugieren que algunos anticuerpos monoclonales específicos de SARS-CoV pueden ser efectivos para neutralizar el 2019-nCoV.
Ciertamente, es tarea de todos contribuir para detener los brotes que han surgido en tan poco tiempo; por ello se aconseja a la población en general que siga todas las recomendaciones para prevenir el contagio, y aunque los números fuera de China son relativamente bajos en comparación con los números dentro de ella, cada país debe estar preparado para cualquier situación relacionada con este virus.
Conflicto de interesesTodos los autores declaran que no existieron conflictos de intereses.







![A) Partícula de coronavirus. Este tipo de virus contiene un material genético de ARN de hebra sencilla de polaridad positiva [(+) ssARN], con un tamaño de 27-32kilobases. Está constituido por una nucleocápside y esta a su vez está compuesta por (+) ssARN y la Nucleoproteína; esta estructura está cubierta de una bicapa lipídica. Aquí se encuentran otras proteínas estructurales del coronavirus como es la proteína Spike que cubre esta partícula viral, así como dímeros de Hemaglutinina-Esterasa (HE); también consta la proteína de Envoltura (E) altamente hidrofóbica y la proteína de Membrana (M), la más abundante en la superficie del virión. B) Organización de los genes en el genoma 2019-nCoV. Los genes estructurales se muestran de color rosa y los genes no estructurales de color azul.](https://static.elsevier.es/multimedia/00142565/0000022100000001/v1_202012300639/S0014256520300928/v1_202012300639/es/main.assets/thumbnail/gr1.jpeg?xkr=veFvTaG4yx3us0l+EPzH7Q==)